
- 연구실 소개
연구실 소개
C1 가스 리파이너리 사업단
UNIST “시스템생물학 및 기계학습 연구실” (김동혁 교수)
연구실 개요
시스템생물학 및 기계학습 연구실(Systems Biology and Machine Learning Lab) 에서는 유기체의 생물학적 현상을 시스템적으로 규명함과 동시에 기계학습을 활용하여 생물화학적 데이터를 다루는 연구를 진행하고 있습니다.
미생물의 전사체 조절 알고리즘을 규명하기 위해 Chromatin ImmunoPrecipitation이나 전사체 분석과 같은 실험 방법을 통해 얻은 데이터를 기반으로 분석함으로써 각 전사 네트워크를 구성하는 방식을 사용하고 있으며, in silico 대사 모델(Genome-scale metabolic models, GEMs)을 활용하여 정해진 반응물 조건에서의 균주 내 대사 흐름과 생성물의 종류와 양 등을 예측하는 방법을 같이 사용하고 있습니다. 이는 실험적으로 규명하기 힘든 다량의 유전체 변화를 시뮬레이션을 통해 빠르고 정확하게 진행할 수 있다는 장점이 있으며, 여러 오믹스 데이터를 통합함으로써 여러 모델 균주들의 대사 조절 네트워크를 재구축하기 위해 연구하고 있습니다.
또한, 최근 각광을 받고 있는 기계학습을 활용하여 대용량 데이터의 복잡도를 낮춤으로써 연계된 시스템생물학/생물정보학 분석을 쉽게 하려는 시도도 병행하고 있습니다. 예를 들면, 머신러닝을 이용해 기존에는 쉽게 분석할 수 없는 단백질 구조의 예측과 같은 생물학적 데이터에 활용하는 연구를 진행 중이며, 소재 데이터 및 화학 공정 데이터에 활용하여 공정 과정의 개선, 조성 분석 및 결과 예측에 적용하는 연구를 수행하고 있습니다. 이를 통해 생명 중심의 연구에서 화학공학, 그리고 머신러닝 쪽으로 점차 연구분야를 넓혀가고 있다.
주요 연구내용 소개
1. 시스템생물학
1) 전사 조절 연구
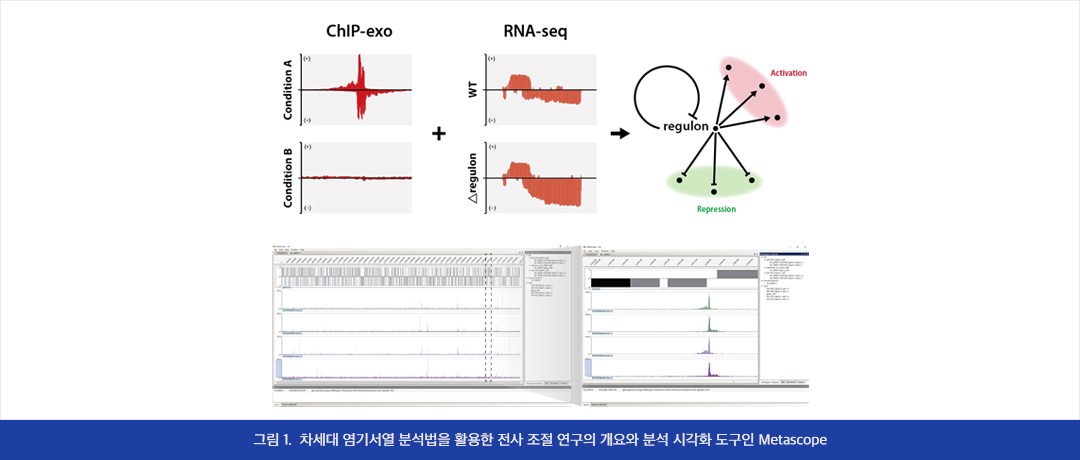
저희 연구실은 박테리아 균주의 전사 조절을 분석하기 위해 차세대 염기서열 분석법 (Next Generation Sequencing, NGS)을 활용하는 전사체 분석 (RNA-seq)과 크로마틴 면역침강법 (ChIP-exo)을 진행하고 있습니다. 또한 실험데이터의 분석을 위해 자체 제작한 분석 파이프라인과 차세대 염기서열 분석법 데이터 시각화 프로그램 소프트웨어 (Metascope)를 활용하여 여러 가지 실험 결과를 통합하여 전사인자에 대한 포괄적인 정보를 확보하고 있습니다. 대상 미생물로는 대장균(E. coli) 및 엔테로박테리아(Salmonella, Klebsiella 등) 뿐만 아니라 산업용 박테리아(Pseudomonas, M. extorquens 등)를 대상으로 전사 조절 연구를 수행하고 있습니다. 대표균주인 대장균 E. coli K-12 MG1655에서는 총 300여개의 전사인자 중 약 10%의 전사인자에 대한 타겟 유전자들을 실험적으로 발굴하고 그 기능을 연구했습니다.
2) 대사 모델 연구
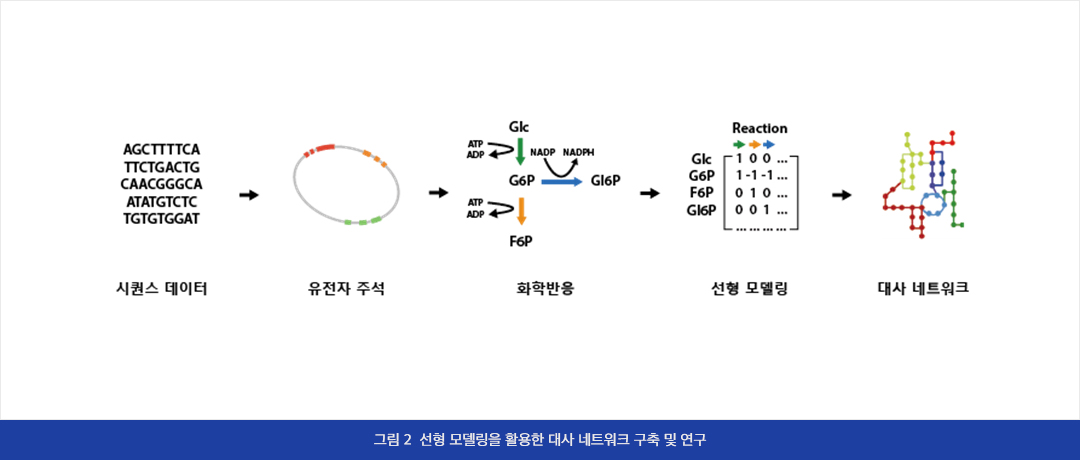
고 처리량 시퀀싱 기술로 읽어진 고품질의 유전체 서열을 기반으로 다양한 데이터베이스를 통해 각 유전자의 주석을 파악함으로써 유전체의 기본 정보를 파악하고 있습니다. 이와 관련된 여러 대사 물질 및 화학 반응을 확인하여 선형 모델링을 기반으로 대사 네트워크를 구성하여 이를 연구에 활용하고 있습니다. 이는 목표 물질에 관련된 생합성 경로를 확인하고 시뮬레이션 함으로써 대사 흐름을 예측할 수 있습니다. 또한 유전자 제거 혹은 유전자 삽입과 같은 조작을 간단히 진행할 수 있으므로 새로운 생합성 경로를 발굴하는 방법에도 유용하게 사용하고 있습니다. 현재는 여러 균주의 대사 모델을 구축하는 것과 함께 이들의 정확도를 높이기 위한 방법을 연구하고 있습니다.
3) C1 생물 전환 박테리아 및 FDH 연구
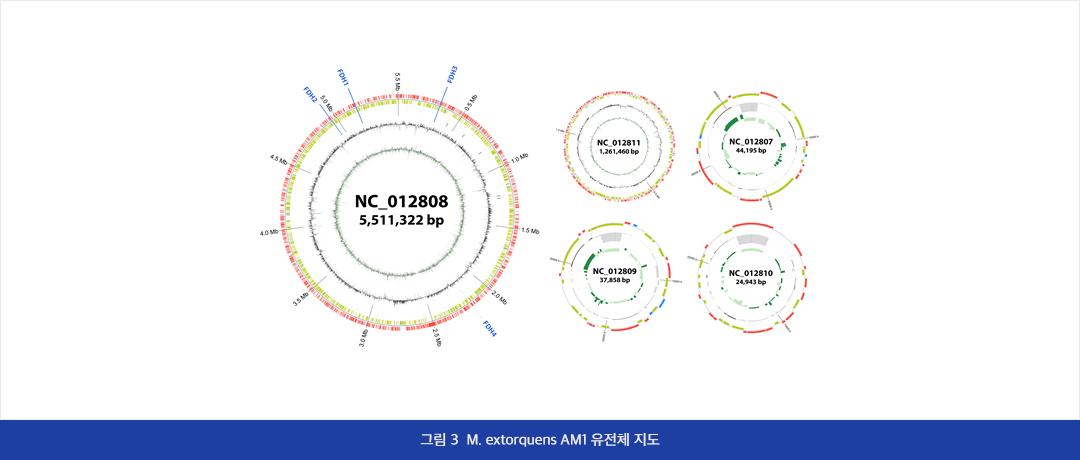
C1 가스 전환 박테리아 중 Methylorubrum extorquens AM1은 산업용으로 각광받고 있지만 아직까지 균주 자체에 대한 연구가 많이 진행되지는 않은 상태입니다. 저희 연구실에서는 이 균주의 유전체를 기반으로 하여 시스템 생물학적 방법을 통해 균주의 특성을 파악하는 연구를 진행하고 있습니다. 이를 위하여 앞서 언급한 전사 조절 연구와 함께 전사체 분석, 대사 모델 구축과 같은 방법을 사용하고 있습니다. 이를 통해 M. extorquens AM1 균주의 기능 유전체학적 지도를 완성하여 목표로 하는 생물 전환을 효율적으로 이루어낼 수 있을 것으로 예상하며, 이를 기반으로 FDH 효소를 시스템생물학적인 관점에서 더 이해하려는 노력을 계속하고 있습니다.
2. 기계학습
1) 단백질 구조 예측
단백질의 아미노산 서열만으로 3차구조를 예측할 수 있는 프로그램인 알파폴드 (Alphafold)가 최근 주목받고 있습니다. 이는 기계학습을 활용하여 수많은 신경망을 통해 수천 개의 단백질을 학습시켜 그 구조를 예측하는 프로그램입니다. 하지만 기계학습의 특성상 학습시킨 단백질과 유사한 단백질에 대해서만 높은 유사도를 보여주기 때문에 이에 대한 범용성을 확보하고자 연구를 진행하고 있습니다.
단백질 구조 예측의 과정으로 특징 추출 과정, 거리 예측 과정, 마지막으로 뒤틀림 각도 예측 과정 이렇게 세 단계로 나눌 수 있으며, 특징 추출의 경우, BLASTP와 HHblits와 같은 MSA (Multiple sequence alignment) 프로그램들을 활용하여 정렬 후 각 잔기별 아미노산의 분포 확률, 유사가능도(pseudolikelihood) 밑 가중치를 통해 재해석된 분포 확률과 같이 여러 가지 아미노산 서열상의 규칙을 확보합니다. 특징 추출은 단백질 구조 예측 파이프라인에서 가장 중요한 부분이고, 자세한 내용이 공개되지 않았기에 수많은 연구자들이 개발하고 있는 부분입니다.
또한 거리 예측과 뒤틀림 각도 예측의 경우, 학습시킨 신경망을 활용하여 데이터를 확보하는 작업을 진행하고, 이를 모델로 구현하는 작업을 위해 PyRosetta라는 프로그램을 활용합니다. 이를 통해 단백질 정보 은행(Protein Data Bank, PDB)형식으로 모델 정보를 획득함으로써 단백질 모델을 얻을 수 있고 PyMOL을 통해 그 모델을 3차원 구조로 가시화할 수 있습니다. 본 연구를 통해 엑스선 결정법(X-ray crystallography) 및 초저온 전자 현미경(cryogenic electron microscopy, cryo-EM)과 같은 복잡한 과정 없이 쉽게 구조를 예측함으로써 그 기능을 유추할 수 있고 더 나아가서 분자동역학 시뮬레이션을 통해 활성과 같은 특성도 분석할 수 있을 것으로 예상하고 있습니다.

2) 화학 공정 데이터 연구
최근 많은 화학 공정에 기계학습을 도입하는 연구가 진행되고 있습니다. 저희 연구실에서는 베이지안 최적화와 생성적 적대 신경망과 같은 여러 가지 방법을 활용하여 소재 및 화학 반응에 적용하는 연구가 진행 중입니다. 그 예로 선형판별분석(LDA, Linear Discriminant Analysis)를 통해 upconversion nanoparticle 촉각센서 필체감정시스템을 구현하였습니다.
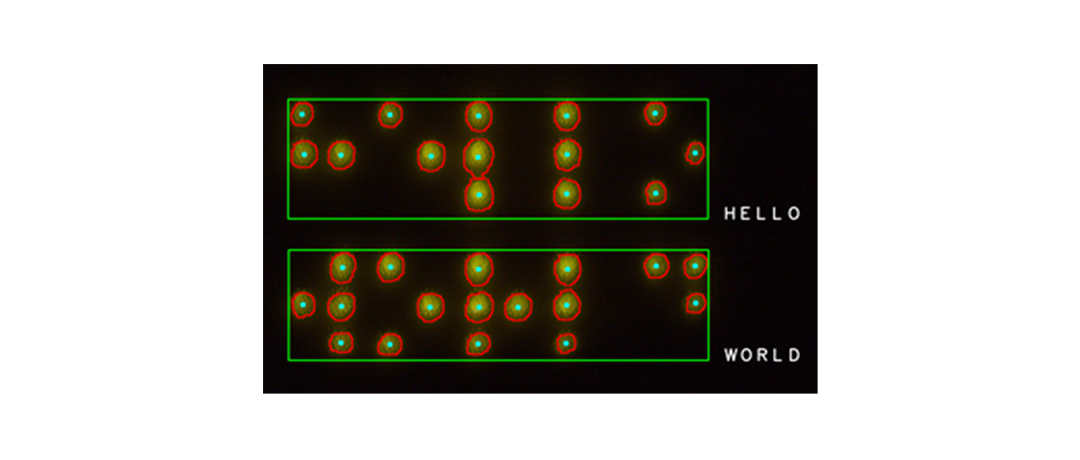
이를 위해, OpenCV 를 이용해 촉각센서 이미지 데이터의 shear force 와 normal force factor 을 추출하여 가장 분류가 잘 되는 축으로 선형판별하였으며, 해당 시스템은 3명의 필체를 성공적으로 분류할 뿐만 아니라 shear force factor을 포함할때 그렇지 않을 때 보다 분류 성능이 높았습니다. 또한, 클러스터링 성능 평가를 통해 본 결과를 정량적으로 확인할 수 있었으며, 이를 통해 새로운 필체 분석 방식을 제안함과 동시에, high spatial resolution 을 특징으로 하는 촉각센서의 경쟁력을 여러 분야에 적용할 수 있는 가능성을 확인할 수 있었습니다. 그 적용 분야 중 하나로 실시간 점자 인식 시스템을 구현하였습니다.
opencv 를 이용해 점자 영역을 추출하고, 이를 좌표화 하여 실시간으로 음성출력하는 본 시스템은 약 500마이크로미터 정도의 작은 점자도 정확하게 변환함으로서 보다 많은 정보를 담을 수 있는 장점이 있습니다.
연구실 현황

시스템생물학 및 기계학습 연구실은 김동혁 교수님을 비롯하여 박사후연구원 1명, 박사과정 학생 2명, 석박사 통합과정 10명으로 총원 14명으로 구성되어 있습니다. 최근 5년간 33편의 논문을 게재하였으며, "C1 가스 리파이너리 사업단“, ”폐플라스틱탄소선순환센터“등 여러 가지 연구 사업에 참여하고 있습니다.


